MARQUAGE SPECIFIQUE DE L’ADN
En cancérologie, la détection
de la cellule pathologique est l’application la plus développée.
Cette détection repose essentiellement sur la mesure d’un
contenu anormal d’ADN dans le noyau de la cellule tumorale. De
très nombreuses études font maintenant appel à la mesure de
l'ADN en cytométrie : dans le domaine végétal, animal, en
parasitologie...
Marquage
spécifique de l’ADN
Il existe de nombreux colorants
de l’ADN pour les études en CMF.
Le Hoechst est un dérivé de benzimidazole qui émet une
fluorescence bleue quand il est excité dans l’UV. Il a une haute
affinité pour l’ADN et se lie préférentiellement aux bases A-T,
mais ce n’est pas un intercalant. Le DAPI présente les mêmes
caractéristiques que le Hoechst et peut être interchangé avec lui.
La mithramycine, la chromomycine sont des antibiotiques excitables
à 445nm et qui émettent à 575 nm (orange). Ils se lient aux
régions G-C par des mécanismes non-intercalants.
L’iodure de propidium et le bromure d’éthidium sont les colorants
les plus utilisés. Ces deux colorants sont des intercalants aussi
bien des doubles brins d’ADN que d’ARN. Tous les deux sont
excitables dans le bleu et émettent dans le rouge (615 nm).
Il existe de très nombreuses autres
molécules permettant de marquer l'ADN avec des caractéristiques
spectrales très différentes permettant d'utiliser de nombreuses
sources d'excitation différentes comme les Sytox (bleu, vert,
orange, rouge), les Vybrant DyeCycle (violet, vert, rouge)...
Certaines ne franchissent pas la
barrière membranaire comme l'IP, le DAPI et les Sytox. Cette
propriété peut être exploitée pour différencier les cellules
vivantes des cellules mortes puisque ces dernières ont perdues
leur inégrité membranaire. Les cellules vivantes ne prennent donc
pas le colorant alors que les cellules mortes deviennent
fluorescentes. Il est aussi possible de perméabiliser celles-ci
afin que le colorant puisse accéder à l'ADN.
D'autres au contraire comme les
Vybrant peuvent diffuser au travers de la membrane cellulaire pour
marquer l'ADN permettant d'obtenir les informations du contenu
d'ADN sur des cellules vivantes.
Stœchiométrie de la coloration
Il est important que la
fluorescence émise par la sonde soit proportionnelle à sa fixation
sur l’ADN. Dans le cas contraire il n’est pas possible de mesurer
un contenu en ADN précis.
Les intercalants, bien que leur fixation soit liée à
l’accessibilité de l’ADN, sont plus utilisés pour mesurer les
contenus en ADN car la teneur en A-T et G-C n’intervient pas dans
leur fixation.
Elimination
des doublets
La fiabilité des résultats
repose sur la possibilité de ne pas prendre en compte les
agrégats cellulaires. En effet, comment distinger un agrégat de
2 ou plusieurs cellules puisqu'au final la quantité d'ADN
mesurée sera le multiple de la quantité d'une cellule unique..
La méthode utilisée repose sur la possibilité d'analyser le
profil du signal recueilli, son aire, sa hauteur ou son temps de
passage (temps de vol, time of flight, TOF, width selon la
machine) (Figure 1).
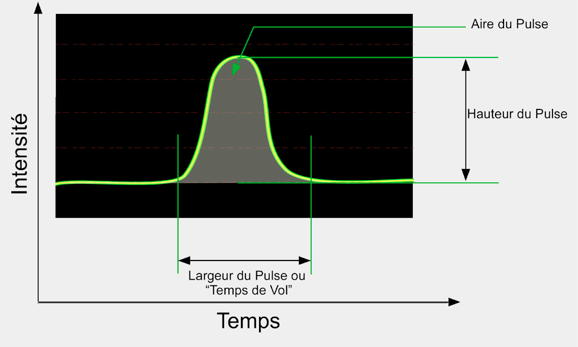 Figure
1 : Profil du signal
Figure
1 : Profil du signal
En utilisant des combinaisons de ces paramètres, il est possible
de distinguer les doublets des singulets (
Figure 2). Cette
méthodologie est aussi utilisée pour le tri cellulaire afin
d'éviter la contamination du tri par des cellules non désirées. En
effet, lors du tri, le cytomètre voit l'agrégat comme une seule
cellule, si cet agrégat est constitué d'une cellule négative et
d'une cellule positive, il sera vu comme une cellule positive. Si
cet agrégat est trié comme tel, il y aura une cellule négative
triée en même temps que la cellule positive aboutissant à une
diminution de la pureté des cellules triées.
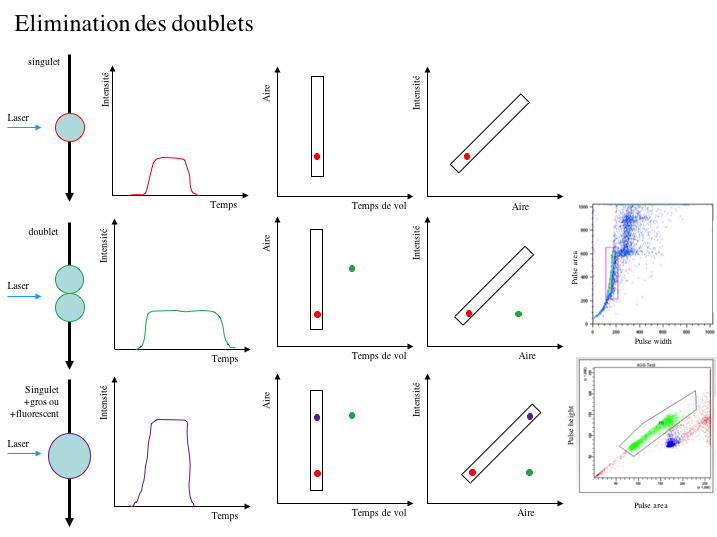 Figure
2 : Principe d'élimination des doublets
Figure
2 : Principe d'élimination des doublets
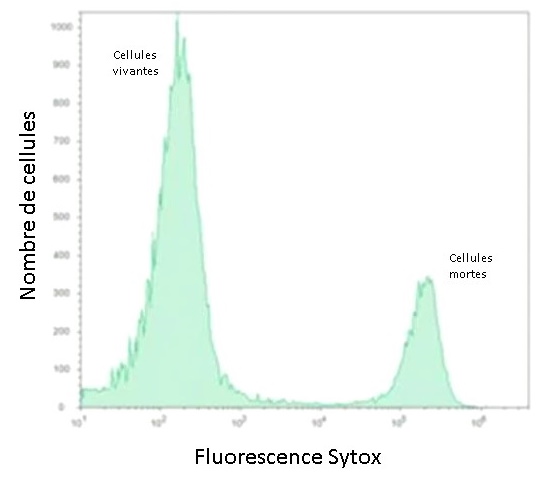
Viabilité cellulaire
L'utilisation de marqueurs de l'ADN ne franchissant pas la
barrière membranaire permet de déterminer très facilement le
pourcentage de cellules vivantes/mortes. Les cellules mortes ayant
des membranes altérées qui laissent passer le colorant deviennent
fluorescentes (Figure 3).
Figure 3 : Marquage des
cellules mortes par le Sytox
Lors de marquages sur des
populations complexes ce type de marquage est important car les
cellules mortes présentent très souvent des marquages non
spécifiques qui peuvent fausser les résultats. Dans le cas du tri
cellulaire, cela évite de trier des cellules mortes si l'on veut
remettre la population triée en culture.
Evaluation
de la quantité d'ADN
En perméabilisant les cellules, le
marquage de l'ADN va permettre de quantifier la quantité présente
au sein de la cellule. Attention toutefois, l'ARN double brin peut
être aussi marqué par les intercalants et s'ajoutera à la mesure
de l'ADN, pour éviter cela il faudra utiliser un tampon de
marquage dégradant cet ARN (RNase). Il faut aussi que
l'accessibilité de l'ADN au colorant soit optimisée. Il existe de
très nombreux protocoles de marquage de l'ADN en fonction des
types cellulaires, il conviendra de tester ses propres cellules
avec plusieurs protocoles afin que la mesure soit optimisée.
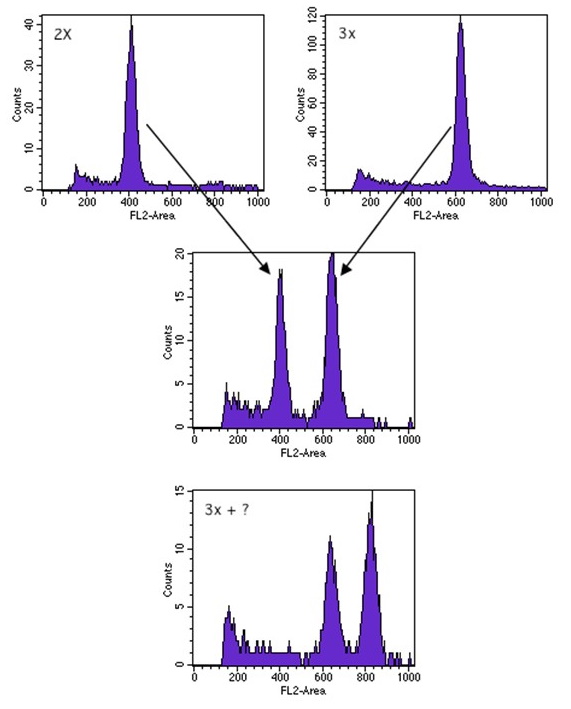
Cette application est utilisée dans
de nombreux domaines comme la recherche agronomique où il a été
constaté par exemple que la quantité d'ADN de certaines plantes
variait en fonction de l'altitude ou de leur situation
géographique (plus l'altitude est élevée, plus il y a d'ADN,
gradient de quantité d'ADN entre les caféiers de l'Afrique de
l'ouest et l'Afrique de l'est) (Figure 4), en pisciculture où on a
constaté que les males triploïdes présentaient de meilleurs
rendements de croissance que leurs congénères diploides d'où la
recherche de conditions d'élevage pour obtenir une majorité de
males triploides (température, pH du milieu)...
Figure 4 : Mesure de la
quantité d'ADN de plans de caféiers. En haut, plan contrôle 2X
et 3X. Au milieu mélange 2X et 3X. En bas mélange contrôle 3X
et échantillon inconnu mesuré à 4X.
Le Cycle cellulaire
“Le cycle cellulaire représente
l’intégralité de la période de division, c’est à dire l’ensemble
des événements biochimiques et morphologiques qui sont
responsables de la prolifération cellulaire”.
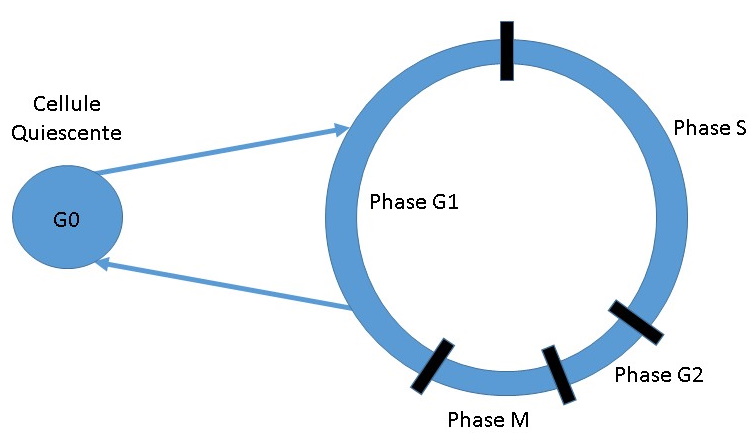
La mesure du cycle cellulaire par des méthodes classiques de CMF
divise le cycle en trois phases: G0/G1, phase d’activation des
cellules, S, phase de synthèse de l’ADN, G2/M phase de mitose (Figure
5 et 6). La distinction entre G0 (phase quiescente) et G1
(phase de préparation à la synthèse d’ADN) ainsi que G2
(préparation de la mitose) et M (mitose) est impossible avec une
méthode utilisant un intercalant comme l’iodure de propidium.
Figure 5 : Les différentes
phases du cycle cellulaire
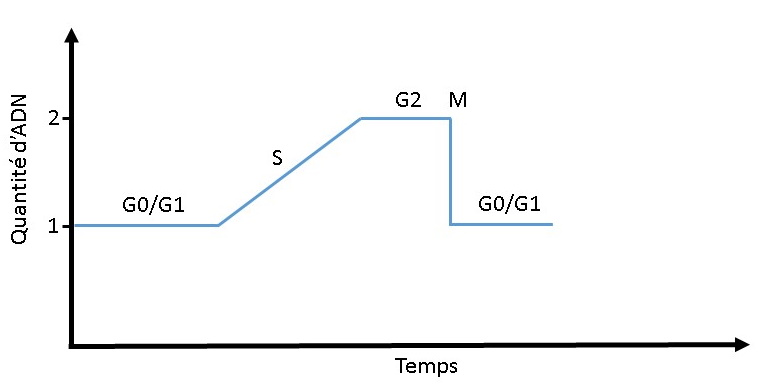
Figure 6 : Evolution de la
quantité d'ADN au cours du cycle
La CMF offre une méthodologie
rapide et simple à mettre en oeuvre pour l’analyse du cycle
cellulaire. Elle permet de suivre la distribution des cellules
dans les différentes phases du cycle en fonction de divers stimuli
ou de l’ajout de certaines drogues. Elle permet aussi de voir la
présence de cellules avec des contenus anormaux d’ADN...
La plupart des applications qui
concernent le cycle cellulaire n’utilisent qu’un seul paramètre,
le contenu en ADN (Figure 7). Des programmes mathématiques
calculent les différentes phases du cycle. Les principales
applications concernent la pharmacologie: étude de l’effet de
drogues sur le cycle cellulaire, la cancérologie: pour déterminer
la prolifération d’une tumeur et voir son contenu en ADN par
rapport aux cellules normales.
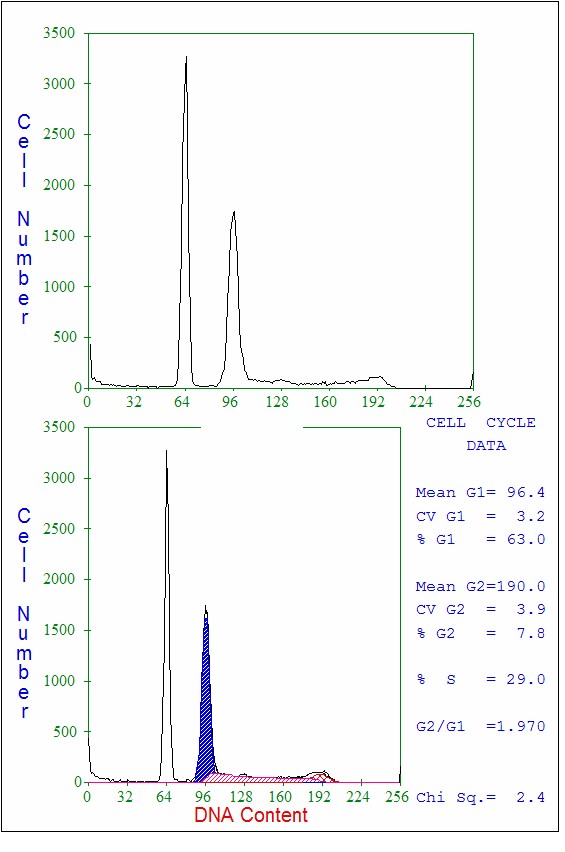 Figure
7 : Analyse du cycle cellulaire d’une lignée tumorale.
Figure
7 : Analyse du cycle cellulaire d’une lignée tumorale.
Il existe d'autres méthodes
d'évaluation du cycle cellulaire et qui permettent de calculer
plus de paramètres du cycle cellulaire. Ces méthodes font appel à
un marquage classique de l'ADN avec un intercalant mais on ajoute
l'incorporation d'analogues de la thymidine. La technique au BrdU
nécessite la mise en culture des cellules avec cet analogue (Figure
8) qui est incorporé lors de la phase de synthèse de l'ADN.
Il faudra ensuite le révéler au moyen d'un anticorps spécifique
capable de pénétrer dans le noyau pour se fixer au BrdU. Cette
méthode nécessitait la dénaturation de l'ADN à l'acide
chlorhydrique ce qui dénaturait les protéines et empéchait de fait
de coupler le marquage du cycle avec un marqueur membranaire. Il
existe maintenant une variante plus douce permettant d'étudier en
plus les marqueurs par immunofluorescence (Carayon et al.). En
jouant sur les temps d'incubation avec l'analogue à la thymidine
on peut calculer des informations sur la cinétique du cycle
cellulaire.
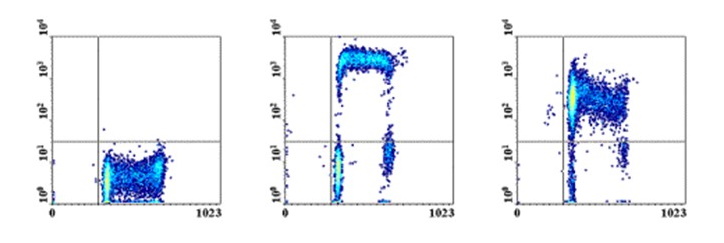
Figure 8 : Marquage du cycle
cellulaire par le BrdU. En X, marquage à l'iodure de
propidium, en Y marquage BrdU. A gauche, simple marquage IP,
au milieu double marquage sur cellules standards, à droite,
double marquage sur cellules bloquées en phase S
De nouvelles méthodes ne
nécessitant pas de dénaturation de l'ADN sont apparues sur le
marché comme la méthode ClickIt de chez ThermoFischer qui fait
appel à un analogue de la thymidine modifié (EDU modifié). La
révélation se fait sans dénaturation car la molécule de révélation
est suffisamment petite pour aller se lier à l'EDUmodifié après
fixation et perméabilisation par un détergent (Figure 9).
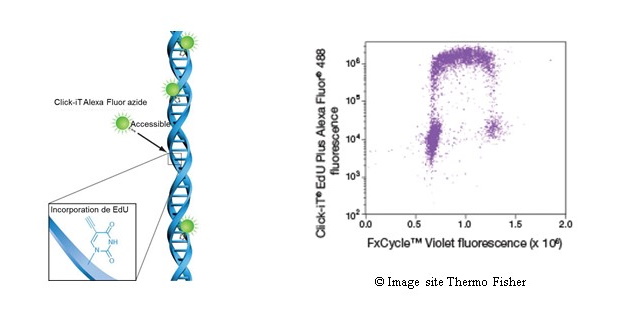
Figure 9: Marquage du cycle
cellulaire par la technique ClickIt. A gauche le principe, à
droite double marquage ClickIt (en Y) avec marqueur de l'ADN
FxCycle (en X).