
AUTRES APPLICATIONS
Les gènes rapporteurs (reporter genes)
Selon la définition Wikipedia : 'Un gène rapporteur est un gène dont le produit (protéine) possède une caractéristique lui permettant d'être observé en laboratoire (fluorescence, activité enzymatique détectable). Les gènes rapporteurs sont utilisés pour permettre de visualiser ou mesurer l'expression d'un gène d'intérêt, pour cela le gène rapporteur peut être fusionné au gène étudié, ou mis sous le contrôle du promoteur de ce dernier'.
En cytomètrie, les gènes rapporteurs sont très utilisés pour la sélection de populations modifiées lors de l'utilisation de trieurs de cellules. L'avantage de cette méthodologie est qu'il n'est pas nécessaire d'utiliser une sélection par un antibiotique. Il existe de très nombreux gènes rapporteurs avec des caractéristiques spectrales différentes comme on peu le voir dans la Figure 1 (Tounsia Aït Slimane, CHU St Antoine). Il est aussi possible d'utiliser plusieurs gènes rapporteurs au sein d'une même expérimentation.


Figure 2 : Comparaison d'une lignée avant et après intégration d'un gène rapporteur fluorecent (GFP).
La quantification de protéines
La cytométrie en flux est une méthode qui fournit des informations relatives quand à la taille, la morphologie et l'intensité d'un marquage. Il est toutefois possible de déterminer avec plus de précision la quantité de protéines présentes à la surface d'une cellule ou circulantes en calibrant l'appareil.
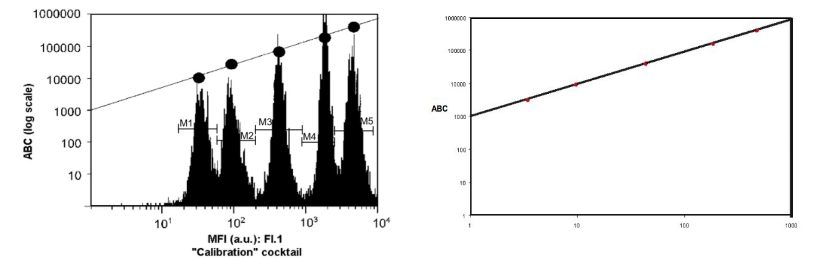
Pour la quantification des antigènes membranaires, il existe par exemple un kit de billes permettant de définir une courbe étalon nécessaire au calcul du nombre d'antigènes présents par cellule (QUIFIKIT commercialisé par Agilent). Le principe consiste à utiliser des billes comportant une quantité d'antigènes connue qui servira à déterminer la courbe étalon. Le kit intègre, en plus d'un tube avec 2 types de billes (négatives et forte densité d'anticorps), cinq populations de billes avec des densités d'anticorps de souris croissantes et connues. La présence de ces anticorps sera révélée par l'utilisation de fragments F(ab')2 de chèvre reconnaissant l'anticorps de souris présent à leur surface. Cet anticorps sera utilisé en conditions saturantes pour révéler les anticorps présents sur les billes mais aussi les anticorps de souris utilisés pour révéler la présence d'antigènes à la surface des cellules testées (Figure 3). Il suffira ensuite de reporter l'intensité moyenne de fluorescence de la population étudiée pour déterminer la densité antigénique de l'antigène mesuré.
Pour la quantification de protéines
circulantes, le principe est à peu près le même (Figure 4).
Il consiste à utiliser des billes qui présentent un anticorps
capable de reconnaitre un épitope de la protéine à analyser ainsi
qu'un anticorps fluorescent capable de reconnaître un second
épitope de cette protéine mais qui n'interfère pas avec le site de
fixation de l'anticorps présent à la surface des billes. La
protéine à quantifier est ainsi captée par la bille et se fixera à
sa surface de façon proportionnelle à sa concentration dans le
milieu étudié et l'anticorps servira à révéler la présence de
cette protéine à la surface de la bille.
Comme avec le QUIFIKIT, en créant une courbe étalon, on sera capable de déterminer avec précision la concentration de la protéine étudiée. En utilisant des billes elles-mêmes fluorescentes en fonction du type de protéines à détecter et en jouant sur l'intensité de fluorescence de ces billes, la société Becton Dickinson est capable de fournir des kits (CBA, ©, Becton Dickinson) susceptibles d'analyser dans le même échantillon jusqu'à 30 protéines différentes à des concentrations jusqu'à seulement 0,274 pg/litre. Un exemple de test CBA est montré dans la Figure 5. L'intérêt de ce type de test est qu'il ne nécessite que de 25 à 50 microlitres d'échantillon à analyser.
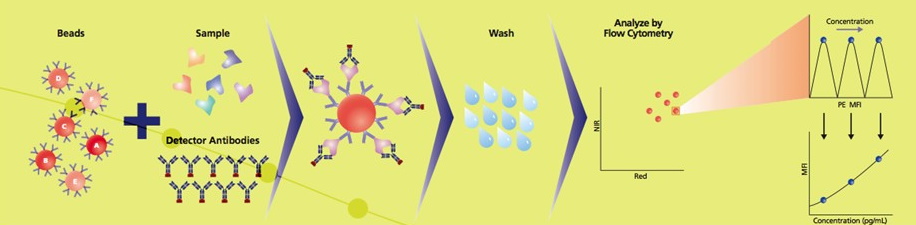
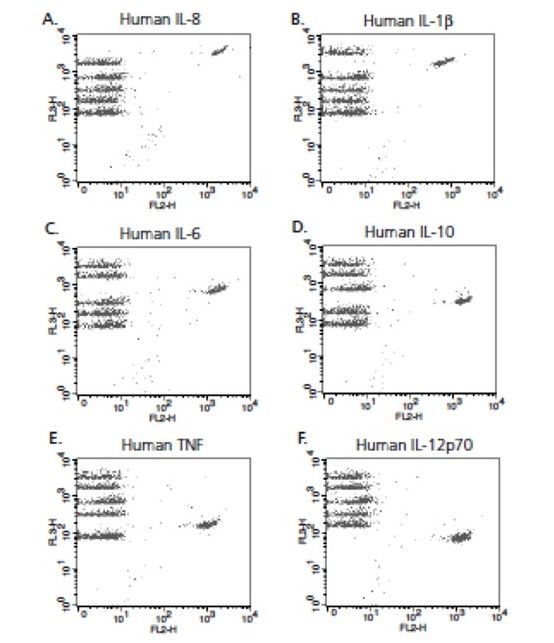
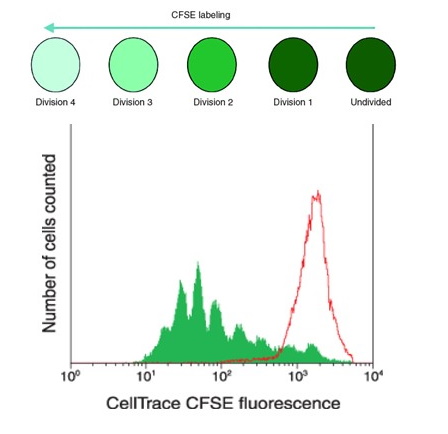
Figure 6 : Principe de mesure de la prolifération cellulaire (en rouge temps=0, en vert courbe après plusieurs divisions)
Analyse des cellules souches
Les cellules souches sont des cellules indiférenciées capables de générer des cellules spécialisées de tous types par différenciation cellulaires. Elles suscitent de grands espoirs en biothérapies et font l'objet de très nombreuses recherches.
La première méthode pour visualiser/séparer ces cellules par cytométrie en flux reposait sur une caractéristique spécifique de ces cellules qui étaient capables d'expulser un colorant(le Hoechst 33342) de leur cytoplasme vers l'extérieur de la cellule. Ce colorant qui diffuse au travers de la membrane cellulaire et marque l'ADN se faisant expulser des cellules souches, celles-ci présentaient un marquage moindre visualisé par cytométrie (Figure 7). Par extension, ces populations de cellules souches prirent le nom de Side Population. L'extériorisation du colorant est un phénomène actif qui peut être inactivé au moyen d'une drogue, le Verapamil. L'ajout de Verapamil inhibant le processus, la population de cellules SP disparait lors de l'analyse par le marquage au Hoechst 33342. Le problème de cette technique est qu'elle nécessitait l'utilisation de lasers UV puissants et n'était donc pas très répandue.
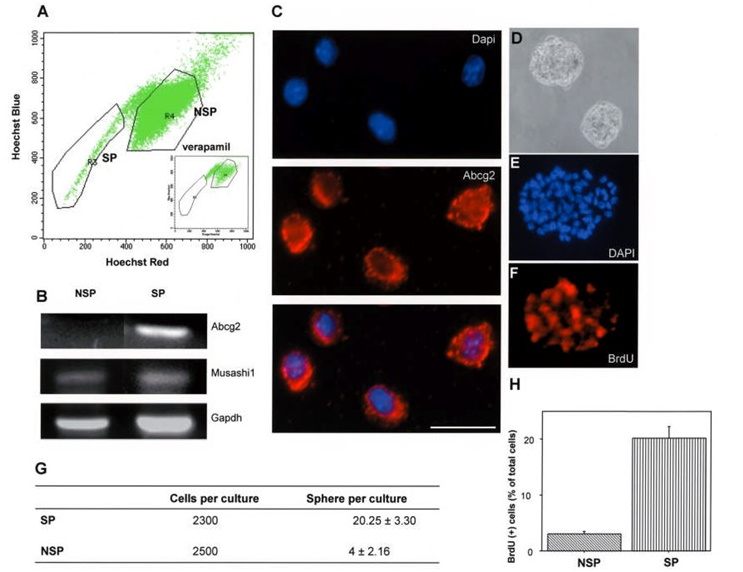
Figure 7 : Analyse des cellules souches (Side Population: SP) par exclusion du Hoechst 33342
Une autre méthode consiste en
l'utilisation de l'activité ALDH (aldéhyde déhydrogénase) pour
isoler ces cellules souches. Il y a une trentaine d'années, une
équipe a découvert que des cellules progénitrices présentaient une
activité ALDH. La mise au point d'un substrat fluorescent
spécifique a permis d'étudier beaucoup plus facilement ces
cellules. Il consiste en une molécule hydrophobe et non-toxique
qui diffuse librement dans les cellules. Une fois dans la
cellules, il est métabolisé dans les cellules ALDH et se retrouve
piégé. Il s'accumule lorsque les systèmes de transport actif sont
neutralisés (principalement par un maintient à 4 degrès). La
fluorescence mesurée est donc proportionnelle à l'activité ALDH.
Le controle négatif est obtenu par l'utilisation de DEAB qui
inhibe l'activité ALDH (Figure 8)
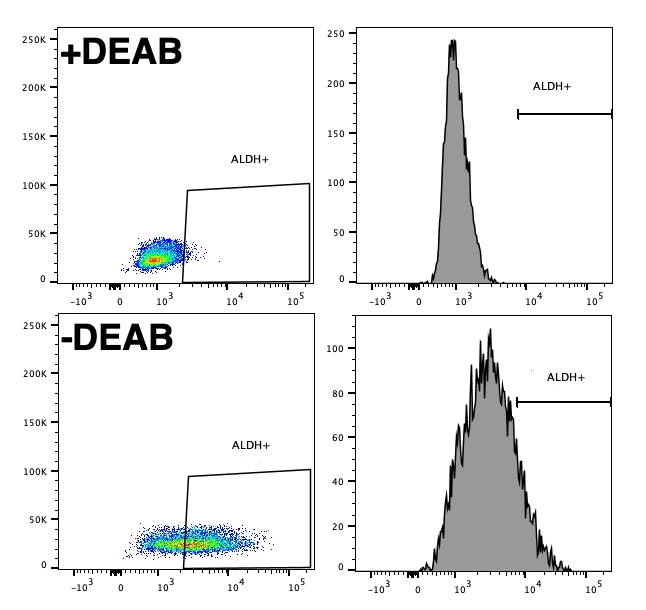
Figure 8 : Marquage de l'activité ALDH sur une lignée cellulaire avec le kit Aldefluor ©